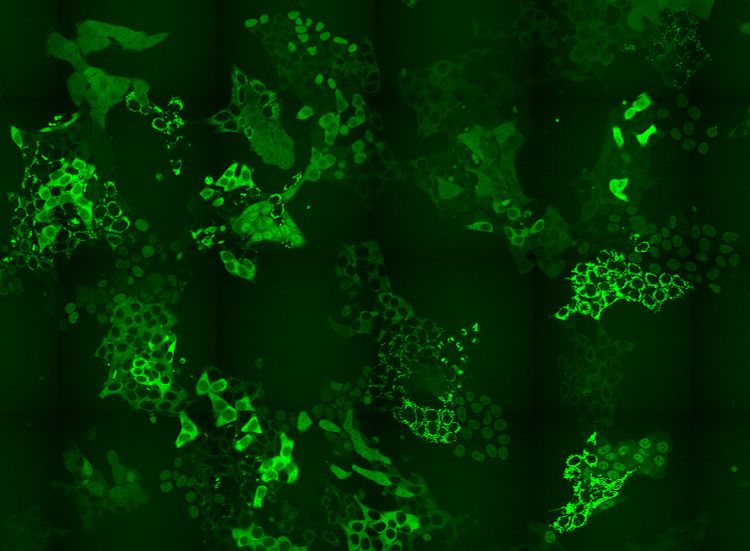
Ein Zellpool mit hunderten Proteinen, die mit der neuen Methode mit fluoreszierenden Markierungen versehen wurden.
Um die Rolle von Proteinen in Zellen zu untersuchen, hängen Forscher derzeit einzelnen Proteinen eine fluoreszierende Markierung an. So lässt sich unter dem Mikroskop der Weg des Proteins in der Zelle und etwa die Wirkung eines Medikaments beobachten. Wiener Forscher berichten nun im Fachjournal "Genome Research" über eine neue Methode, mit der sich erstmals Hunderte Proteine parallel beobachten lassen.
Proteine sind große Moleküle in der Zelle, die für Struktur und Funktion von Gewebe und Organen im Körper erforderlich sind. Sie sind für fast alle Aufgaben des zellulären Lebens verantwortlich und können so vielfältig sein wie die Funktionen, die sie erfüllen. Die Mengen und Lokalisierungen von Proteinen innerhalb der Zelle steuern wichtige Aspekte vieler zellulärer Prozesse und können als Ziele für die Entwicklung von Medikamenten fungieren.
Hochskalierbare Methode
Traditionell verwenden Wissenschafter eine Fluoreszenzmarkierung, um einzelne Proteine zu markieren und ihre Rolle in der Zelle zu untersuchen. Diese Methode erlaubt es, zum Beispiel die Effekte verschiedener Medikamentendosierungen auf ein einzelnes Protein zeitaufgelöst zu untersuchen.
Andreas Reicher und Anna Koren aus der Gruppe von CeMM-Principal Investigator Stefan Kubicek haben eine neuartige hochskalierbare Methode entwickelt, die es zum ersten Mal erlaubt, Veränderungen in einer sehr hohen Anzahl von Proteinen parallel zu beobachten und zu charakterisieren. Mit dieser Methode können nicht nur die Wirkungen bestimmter bekannter Medikamente in den Zellen beschrieben und besser verstanden werden, sondern auch neue Wirkstoffe entdeckt werden, die durch Beeinflussung der Proteinspiegel oder -lokalisationen in den Zellen wirken.
Hunderte von markierten Proteine
Die Forscher entwickelten eine CRISPR-Cas9 basierende Intron-Markierungsstrategie, um Zellpools zu erzeugen, die Hunderte von markierten Proteinen enthielten, wobei in jeder Zelle ein anderes Protein mit fluoreszierenden Markern versehen wurde. Diese Zellpools wurden nun niedermolekularen Substanzen wie einem Molekül zum chemischen Abbau von BRD4 sowie dem zugelassenen Krebsmedikament Methotrexat ausgesetzt.
Die Forscher beobachteten dann mithilfe der Zeitraffermikroskopie, ob es als Reaktion auf die angewandte Behandlung zu Veränderungen in der Konzentration oder subzellulären Lokalisierung der markierten Proteine in irgendeiner der Zellen im Pool kam. Die von ihnen angewandte Markierungsstrategie ermöglichte es ihnen dann, mithilfe von In-situ-Sequenzierung festzustellen, welche der hunderten Proteine im Zellpool die Lokalisierung veränderten. Auf diese Weise konnten sie sowohl erwartete als auch bisher unbekannte Auswirkungen dieser Substanzen auf die Abundanz und Lokalisation einzelner Protein feststellen.
Wirkstoffe schneller entwickeln
"Unsere Studie beschreibt eine Technologie, die nicht nur zum ersten Mal Intron-Markierung auf einen Genpool anwendet, sondern auch in allen drei Schritten – Intron-Markierung, zelluläre Bildgebung und In-situ-Sequenzierung – deutlich optimiert wurde. Diese Methode kann auf chemische Bibliotheken und Kandidatenmoleküle angewandt werden, um Wirkstoffe zu entwickeln und umfassend zu charakterisieren. Dadurch kann die Entwicklung von Medikamenten mit neuartigen Wirkmechanismen, wie der Induktion des chemischen Proteinabbaus und der Veränderung von Protein-Protein-Interaktionen, beschleunigt werden. Zudem wird die neue Strategie auch großen Einfluss auf die Untersuchung der globalen und subzellulären Proteomdynamik haben", erklärt Kubicek. (red, 19.11.2020)