Seit das britische Unternehmen Deep Mind Technologies 2014 von Google übernommen wurde, hat sich eine Menge getan. Das erklärte Ziel der Firma ist eine flexible künstliche Intelligenz mit multiplen Einsatzgebieten. Im Unterschied zu vergleichbaren AIs setzt Deep Mind nicht ausschließlich auf ein neuronales Netz, sondern ergänzt dieses um einen Kurzzeitspeicher, was ein künstliches Gedächtnis simuliert. Erstmals ins Rampenlicht trat Deep Mind 2016, als die KI Alpha Go den zu diesem Zeitpunkt weltbesten Go-Spieler Lee Sedol in dem komplexen Brettspiel in vier von fünf Spielen schlug. Der Gesamtsieg des Programms gilt als bedeutender Fortschritt in der Entwicklung selbstlernender Maschinen, nachdem Go mit seinen vielen möglichen Spielzügen bis dahin für zu komplex für Computer gehalten worden war.
Meisterhafter Spieler und Lippenleser
2019 hängte der 18-fache Go-Weltmeister Lee seine Spielerkarriere übrigens an den Nagel. Laut einem Interview mit der südkoreanischen Presseagentur Yonhap habe er eingesehen, dass die KI die Oberhand hat und nicht mehr zu schlagen sei. Alpha Gos Nachfolger Alpha Zero war noch höher entwickelt: Während Ersterer die Züge von menschlichen Top-Spielern mehrere Monate lang analysieren musste, um sein hohes Niveau zu erreichen, hatte das Deep-Mind-Team nun einen anderen Weg beschritten: Alpha Zero erlernte Go, Schach und das japanische Spiel Shogi ohne Anschauungsmaterial, indem es sich in hunderttausenden Partien gegen sich selbst zur Meisterschaft trainierte. Allein die Regeln der Spiele waren der KI zu Beginn bekanntgegeben worden.
Bild nicht mehr verfügbar.
So soll Alpha Zero 2017 dem damals weltbesten Schachprogramm Stockfish 8 bereits nach einer Lernzeit von nur vier Stunden weit überlegen gewesen sein. Von 100 Partien verlor Zero keine einzige. Ähnlich flott war die Software bei Go: Nach acht Stunden Training schlug Alpha Zero seien Vorgänger Alpha Go in 60 von 100 Spielen. Aber auch abseits von Spielen bewährten sich die künstlichen Intelligenzen von Deep Mind in herausragender Weise. Nach dem Konsum von 5.000 Stunden Fernsehen war beispielsweise ein entsprechendes Programm deutlich treffsicherer beim Lippenlesen als menschliche Spezialisten. Und auch bei der Simulierung menschlicher Sprache hat Google die Nase vorne: Eine Kombination zweier AI-Programme, von denen eines ebenfalls von Deep Mind stammt, lieferte gesprochenen Text, der von Aufnahmen echter Menschen kaum noch zu unterscheiden ist.
Erfolgreich in wissenschaftlichen Gefilden
All das nimmt sich freilich als Spielerei aus neben jenem Durchbruch, der Googles AI-Schmiede nun gelungen sein soll: Der von Deep Mind entwickelte Algorithmus Alpha Fold 2.0 will das jahrzehntealte Problem der Proteinfaltung gelöst haben. Immerhin berichten "Nature" und "Science" von einem "gigantischen Sprung nach vorne".
Proteine, also Eiweiße, bestehen aus einer Kette von aneinandergereihten Aminosäuren. Die Funktion eines Proteins in einer Zelle hängt allerdings nicht nur von seiner chemischen Struktur ab, sondern vor allem davon, wie diese Kette gefaltet ist. Für die Erkenntnis, welche Struktur sich aus einer Proteinsequenz zumindest theoretisch ergibt, wurde der US-amerikanische Biochemiker Christian Anfinsens 1972 mit dem Nobelpreis in Chemie ausgezeichnet. Kennt man den dreidimensionalen Aufbau eines Proteins, lassen sich daraus wertvolle Schlüsse über dessen Verhalten und Wirkungen ziehen.
Komplexe Proteinfaltung
Doch bis heute stellen die komplexen Proteinstrukturen für die Wissenschafter ein nur sehr aufwendig lösbares Problem dar, an dem sie sich bisweilen die Zähne ausbeißen. Die Zahl möglicher Varianten, wie ein bestimmtes Protein gefaltet sein kann, liegt immerhin bei 10 hoch 300, wie Fachleute schätzen. Auch wenn man einen großen Teil davon von vornherein ausschließen kann, bleiben immer noch genug übrig, um Strukturbiologen und Bioinformatiker jahrelang zu beschäftigen.
Daher gründeten 1994 die Wissenschafter John Moult und Krzysztof Fidelis das Critical Assessment of Protein Structure Prediction (CASP). Das alle zwei Jahre stattfindende Gemeinschaftsexperiment für die Vorhersage der Proteinfaltung hat das Ziel, die besten Methoden für die Prognose von Proteinstrukturen zu finden. CASP liefert Forschergruppen die Grundlage, die Qualität ihrer Methoden zur Vorhersage von Proteinstrukturen ausgehend von der Primärstruktur zu testen und sich einen Überblick über den aktuellen Stand in diesem Forschungsgebiet zu verschaffen.
Bereits 2018 erfolgreich
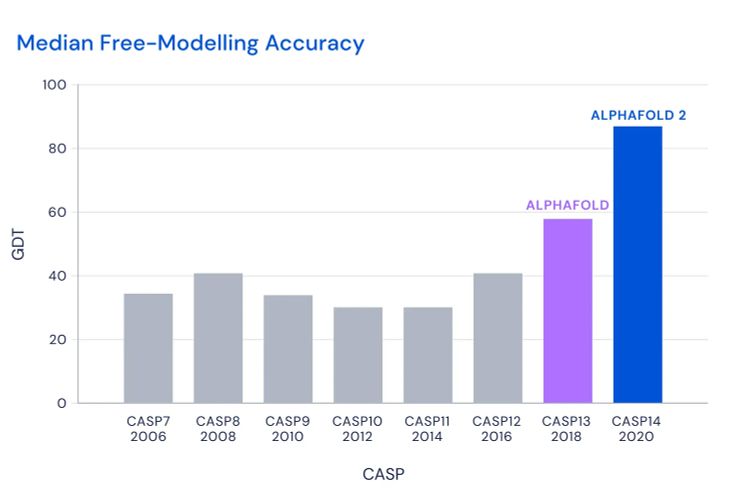
Bei diesem Wettbewerb kommen noch unveröffentlichte Proteinstrukturen zum Einsatz, die bereits von anderen Forschern empirisch aufgedeckt worden waren. Auf diese Weise kann CASP die Vorhersagen der Teilnehmer auf ihre Richtigkeit überprüfen. 2018 nahm auch Alpha Fold an dem Wettbewerb teil. Zu diesem Zeitpunkt war der Algorithmus erst zwei Jahre alt – und doch belegte er unter den 98 Teilnehmern den ersten Platz, indem er bei 25 von 43 Proteinen die beste Struktur vorhersagte. Das Team, das es auf den zweiten Platz schaffte, prognostizierte dagegen die Faltung von nur drei Proteinen.
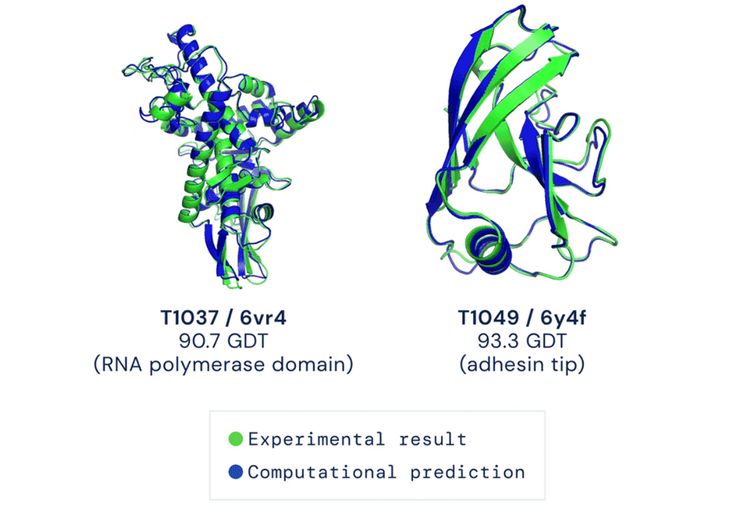
Am jüngsten CASP14-Wettbewerb beteiligte sich die weiterentwickelte Version Alpha Fold 2.0. Der Erfolg der Software war enorm und kann durchaus als Durchbruch gewertet werden: Von knapp 100 Proteinsequenzen sagte Alpha Fold bei 70 Proteinen jene Faltung vorher, die auch in den experimentellen Verfahren eruiert wurde – für die Wissenschafter geradezu eine Revolution in der Proteinforschung. "Alpha Fold hat sich als fähig erwiesen, die Struktur vieler Proteine mit der Genauigkeit teurer und zeitaufwendiger Laborexperimente zu bestimmen", berichten die CASP-Forscher auf ihrer Website.
"Leistungsfähig genug für wissenschaftliche Probleme"
Demis Hassabis, ein Mitgründer von Deep Mind, bezeichnete den Erfolg als "Schlüsselmoment", bei dem sein Unternehmen erstmals beweisen konnte, dass AI "grundlegende, sehr wichtige und reale wissenschaftliche Probleme lösen" könne. "Diese Algorithmen sind mittlerweile leistungsfähig genug, um sie bei wissenschaftlichen Problemen einzusetzen. Nach vierjähriger Entwicklungszeit haben wir nun ein System, das präzise genug ist, um tatsächlich eine Bedeutung und Relevanz für Biologen und Biochemiker zu haben."
Eines der Kriterien beim CASP-Wettbewerb ist der sogenannte Global-Distance-Test-Wert (GDT), der zwischen 1 und 100 liegt. Je höher dieser Wert ist, desto näher wurde die Positionen der einzelnen Aminosäurereste eines Proteins im Vergleich zur tatsächlichen Struktur bestimmt. Im Durchschnitt lagen die Vorhersagen von Alpha Fold 2.0 bei 92,4 GDT, wobei ein Wert ab 90 bereits als vergleichbar mit der Präzision experimenteller Methoden gilt. Bei außerordentlich herausfordernden Proteinen (sogenannte "Hard to Predict"-Proteine) lag der Schnitt sogar bei etwa 87 GDT. Mehr noch: Bei etwa einem Dutzend der "Hard to Predict"-Proteine konnte Alpha Fold 2.0 den Wissenschaftern helfen, die Resultate ihrer Experimente zu optimieren.
Forscher: "Enorme Bereicherung"
Deep Mind will demnächst ein Methodenpaper veröffentlichen und hat angekündigt, mit dem Vorhersagemodell eine beschleunigte Proteinstrukturanalyse für Forschende mit berechtigtem Interesse ermöglichen zu wollen. Auch wenn Alpha Fold bisher noch keine Strukturen von Multidomain-Proteinen oder Proteinkomplexen vorhersagen kann, dürfte die optimierte KI die Forschung und Pharmaentwicklung revolutionieren. Auch bei der Erforschung des Coronavirus hat sich Alpha Fold nützlich gemacht: Anfang des Jahres sagte die Software sechs bis dahin unbekannte Proteinstrukturen von Sars-CoV-2 vorher. Zwei dieser Vorhersagen – ORF3a und ORF8 – sind mittlerweile im Labor bestätigt worden. Das Unternehmen Deep Mind sucht bereits nach Möglichkeiten, Wissenschaftern den Zugang zum Alpha-Fold-System auf "skalierbare Weise" zu ermöglichen, erklärt Hassabis.
Für Wissenschafter ist Alpha Fold eine enorme Bereicherung: "Die Bestimmung einer einzelnen Proteinstruktur erforderte oft jahrelange experimentelle Anstrengungen", sagte Janet Thornton, Strukturbiologin am European Bioinformatics Institute in Hinxton, Großbritannien, und eine der Pioniere bei der Verwendung rechnerischer Ansätze zum Verständnis der Proteinstruktur. "Ein besseres Verständnis der Proteinstrukturen und die Fähigkeit, sie mithilfe eines Computers vorherzusagen, bedeutet ein besseres Verständnis des Lebens, der Evolution und natürlich der menschlichen Gesundheit und Krankheit." (tberg, red, 2.12.2020)